Dependencia con la temperatura del radio hidrodinámico de una proteína intrínsecamente desordenada medida en la ultracentrífuga analítica Optima AUC.
Tipo de contenido: Nota de aplicación
Lance R. English | Department of Chemistry and Biochemistry, Texas State University, San Marcos, Texas, USA
Aysha Demeler | Department of Chemistry, Bergische Universität Wuppertal, Wuppertal, North Rhine-Westphalia, Germany
Steven T. Whitten | Department of Chemistry and Biochemistry, Texas State University, San Marcos, Texas, USA
Borries Demeler | Department of Biochemistry and Structural Biology, University of Texas Health San Antonio, San Antonio, Texas, USA
Resumen
Se realizaron experimentos de ultracentrifugado analítico en un amplio intervalo de temperaturas para medir el radio hidrodinámico de una proteína intrínsecamente desordenada. Aprovechando el control de temperatura altamente estable ofrecido a través de la modificación de la cubierta disponible en la nueva Optima AUC, los experimentos de réplica demostraron parámetros hidrodinámicos altamente reproducibles que sugieren cambios leves en el radio hidrodinámico de una proteína intrínsecamente desordenada y un impresionante rendimiento tanto en control de temperatura como en calidad de los datos de la Optima AUC. Un análisis de datos extensivo con Ultrascan 4.0 identificó pequeñas cantidades de contaminantes en la preparación de las proteínas y proporcionó validación estadística de los resultados experimentales a través de un ajuste global y del análisis de Monte Carlo. Además de los coeficientes de sedimentación y difusión, se determinaron el volumen específico parcial, el radio hidrodinámico, la relación de fricción y los coeficientes de fricción.
Introducción
Una construcción de secuencia invertida de la nucleasa estafilocócica (retro-nucleasa) representa una secuencia de proteínas sin estructura ni función intrínseca,1 y se utilizó en este estudio como sistema modelo para una proteína intrínsecamente desordenada. No se observó colapso hidrofóbico y la estructura de la retro-nucleasa se parece a la de las proteínas biológicas intrínsecamente desordenadas. Para evaluar el grado de trastorno, el radio hidrodinámico puede medirse con técnicas que producen el coeficiente de difusión, que es inversamente proporcional al coeficiente de fricción de la molécula, y el radio hidrodinámico. Estas técnicas incluyen la dispersión de luz dinámica, el fraccionamiento del flujo de campo, la espectroscopía de correlación de fluorescencia y el ultracentrifugado analítico. Aquí, los radios hidrodinámicos de la proteína de la retro-nucleasa se midieron mediante experimentos de ultracentrifugado analítico (AUC) y de velocidad de sedimentación (SV) en función de la temperatura. La reciente llegada de la ultracentrífuga analítica Optima AUC de Beckman-Coulter ofrece importantes mejoras tecnológicas sobre su predecesora, el modelo ProteomeLab XLA/XLI de Beckman-Coulter. Estas mejoras incluyen mayor resolución y precisión radial, mayores velocidades de escaneo, un monocromador muy preciso y, con la reciente actualización de la cubierta de la cámara interna, un control de temperatura increíblemente estable. Mostramos cómo se pueden aprovechar estas mejoras técnicas en los experimentos de SV realizados en la Optima AUC, en combinación con el análisis de datos Ultrascan para obtener resultados altamente reproducibles, para medir con exactitud incluso los cambios más pequeños de los radios hidrodinámicos en función de los cambios de temperatura.
Los experimentos de SV miden el coeficiente de sedimentación, s, y el coeficiente de difusión, D, de un soluto en solución a través de la monitorización de estos dos procesos de transporte en un campo de fuerza centrífuga.
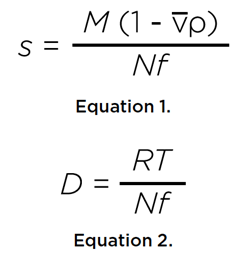
donde M es la masa molar del soluto, v su volumen parcial específico, ρ la densidad del disolvente, R es la constante universal del gas, T es la temperatura en grados Kelvin, y N es el número de Avogadro. Ambos coeficientes son inversamente proporcionales al coeficiente de fricción, f, del soluto, que se relaciona con el radio hidrodinámico rh del soluto a través de la relación Stokes-Einstein:
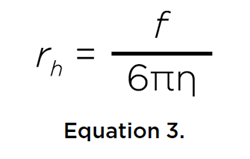
donde η es la viscosidad del disolvente. Además, si se conoce la masa molar del soluto, ya sea por secuencia o por espectrometría de masas, se puede estimar el volumen específico parcial del soluto a partir de estas dos propiedades de transporte:
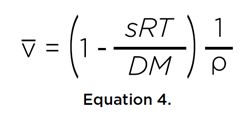
Si el soluto fuese una esfera perfecta con el mismo volumen, podemos derivar el radio, r0, y el coeficiente de fricción, f0, de la esfera mínima:
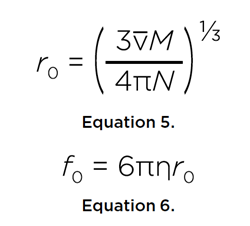
Esto proporciona la relación de fricción o anisotropía, f/f0, del soluto. Una anisotropía de 1,0 indicaría una esfera perfecta, mientras que se esperaría que una proteína intrínsecamente desordenada tuviese una anisotropía de entre 1,5 y 2,5.
Métodos
Experimental
La absorbancia de una solución concentrada de la proteína de retro-nucleasa se ajustó a una densidad óptica de 0,6 unidades de absorbancia a 230 nm mediante dilución con 10 mM de tampón de fosfato, pH 7,0, conteniendo 100 mM de NaCl. Las muestras replicadas se midieron cargando ambos sectores de las piezas centrales de epon estándar de 2 sectores (Beckman-Coulter). Todos los experimentos se realizaron a 50 000 rpm; después de 1 hora de equilibrado de temperatura del rotor en reposo, las temperaturas se fijaron en 4, 12, 20, 28 y 37 °C; los datos se recopilaron mediante detección de intensidad de UV a 230 nm. Se realizaron experimentos de sedimentación durante al menos 800 minutos, o hasta que se recogiesen 1500 escaneos, escaneando cada sector cada 20 segundos en una ultracentrífuga analítica Optima AUC de Beckman-Coulter en el Centro de Ultracentrifugación Analítica de Conjuntos Macromoleculares en el Centro de Ciencias de la Salud de la Universidad de Texas, en San Antonio, utilizando un rotor An60Ti. Todos los datos se analizaron con el UltraScan- III ver. 4.0, lanzamiento 2420.2, 3 Las correcciones hidrodinámicas de densidad y viscosidad del tampón realizadas con el UltraScan se estimaron en 1,00362 g/ml y 1,00866 cP a 20 °C. Los ajustes de viscosidad y densidad para la temperatura del disolvente se realizaron de manera automática con el UltraScan en función del trabajo de Kell.4 El transporte de la sedimentación y la difusión en la celda de ultracentrifugación, se describen a través de la ecuación de Lamm, que puede resolverse utilizando métodos de elementos finitos adaptivos5, 6 (ASTFEM). Los datos de los 1500 escaneos obtenidos de cada sector del experimento de SV se ajustaron mediante combinaciones lineales de soluciones ASTFEM utilizando rutinas de optimización avanzadas 7, 8, 9 que se implementaron en un superordenador.10Para la proteína de retro-nucleasa, obtuvimos la masa molar de la secuencia (16,819 kDa) y usamos este valor para el cálculo de los parámetros hidrodinámicos distintos a los análisis de los datos s, D, y Rh
Los datos de SV experimentales se procesaron previamente mediante análisis espectral bidimensional (2DSA)7 con la eliminación simultánea de las contribuciones de ruido con invariación temporal y radial11 para el intervalo de valores s entre 0,5 y 5 s y la tasa de fricción en un intervalo de entre 1 y 4. A continuación, utilizando los mismos intervalos, se ajustó la posición del menisco como se describe en.12 Los datos resultantes se refinaron aún más mediante regularización parsimoniosa utilizando algoritmos genéticos.8 Para obtener estadísticas de error, se asoció el análisis de Monte Carlo con algoritmos genéticos en el refinamiento final.9 Los resultados del análisis de Montecarlo de muestras duplicadas se combinó y promedió en peso. Los cálculos son de computación intensiva y se realizan en plataformas informáticas de alto rendimiento.11 Todos los cálculos se realizaron en el clúster Lonestar-5 del Centro de Computación Avanzada de Texas, de la Universidad de Texas, en Austin, y en Comet del Centro de Supercomputación de San Diego.
Resultados
Todos los experimentos mostraron un grado muy alto de reproducibilidad (consulte la Figura 1 para un ejemplo a 20 °C), con intervalos de confianza estrechos del 95 % estrecho (= 2σ, consulte la Figura 2). El Optima AUC mostró una impresionante estabilidad a alta temperatura en todos los ajustes de temperatura entre 4 °C y 37 °C, según se determinó a partir del trazado de estado-tiempo registrado en la Optima AUC con incrementos de 1 segundo (Figura 3), con una fluctuación de aproximadamente ±0,1 °C, excepto en la parte temprana del experimento, donde se ve el estiramiento por refrigeración adiabática del rotor, lo que provoca un efecto de enfriamiento de -0,6 °C al acelerar desde el reposo a 50 000 rpm en un rotor AN60Ti. Pero la lógica de control de temperatura devuelve rápidamente la temperatura a los valores establecidos en todos los casos, con un pico máximo de 0,2 °C (0,3 °C a 4 °C) que se amortigua a los valores establecidos en los primeros 20-40 minutos del experimento.
Los parámetros hidrodinámicos obtenidos a partir de los experimentos de velocidad realizados entre 4 °C y 37 °C indicaron solo un leve cambio en todos los valores (consulte la Figura 3 y la Tabla 1). El radio hidrodinámico, rh, aparenta sufrir un ligero aumento a medida que aumenta la temperatura, lo que sugiere más desdoblamiento a temperaturas más altas o una molécula más flexible que puede habitar en un espacio conformacional adicional. El volumen específico parcial mostró una desviación opuesta y consistente a valores inferiores con temperaturas más altas, lo que sugiere menos hidratación a temperaturas más altas, debido posiblemente al desdoblamiento, exponiendo las regiones hidrófobas en la molécula. El radio hidrodinámico y el volumen específico parcial, juntos, contribuyen a un leve aumento de la anisotropía con temperaturas en aumento, con relaciones de fricción que aumentan de 1,96 a 2,11. En resumen, estos resultados demuestran que la Optima AUC proporciona un control de temperatura muy estable y respuestas altamente reproducibles, lo que permite a los investigadores detectar incluso cambios sutiles en los parámetros hidrodinámicos de una pequeña proteína intrínsecamente desordenada.
| T (°C) | s (s x 10-13) | D (cm2/s x 10-7) | rh (nm) | f/f0 | v (ml/g) |
| 37 | 1,29 (1,27, 1,31) | 6,52 (5,96, 7,07) | 3,48 (3,21, 3,80) | 2,08 (1,90, 2,30) | 0,6973 (0,6743, 0,7166) |
| 28 | 1,23 (1,18, 1,28) | 6,26 (6,02, 6,50) | 3,52 (3,39, 3,66) | 2,10 (2,01, 2,18) | 0,7087 (0,7086, 0,7088) |
| 20 | 1,28 (1,02, 1,55) | 6,25 (4,85, 7,65) | 3,43 (2,80, 4,42) | 2,04 (1,67, 2,65) | 0,7037 (0,6967, 0,7081) |
| 12 | 1,27 (1,24, 1,30) | 6,28 (5,38, 7,18) | 3,31 (2,90, 3,87) | 1,97 (1,70, 2,34) | 0,7162 (0,6770, 0,7457) |
| 4 | 1,22 (1,11; 1,34) | 6,10 (5,24, 6,56) | 3,32 (3,04, 3,66) | 1,96 (1,95, 1,98) | 0,7264 (0,7252, 0,7274) |
| Tabla 1: Resultados hidrodinámicos de mediciones de SV o retro-nucleasa a temperaturas en el intervalo de 4 a 37 °C. Los valores entre paréntesis representan intervalos de confianza del 95 % obtenidos en el análisis de Monte Carlo (2 σ). | |||||
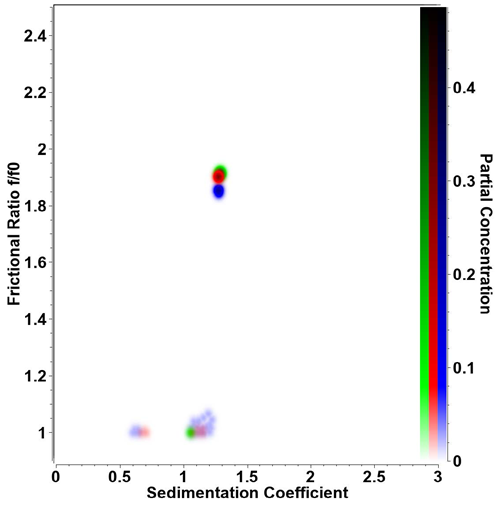 |
| Figura 1. Superposición de un gráfico pseudo-3D de muestras de retro-nucleasa por triplicado medidas a 20 °C. En la Optima AUC se obtiene una reproducibilidad considerablemente alta tanto del coeficiente de sedimentación como de la relación de fricción para experimentos independientes. Incluso los contaminantes de baja concentración (s ≈ 0,65 s y s ≈ 1,1 s) se detectan de forma reproducible en la misma posición en el espacio de parámetros bidimensional. La intensidad del color refleja la concentración parcial. |
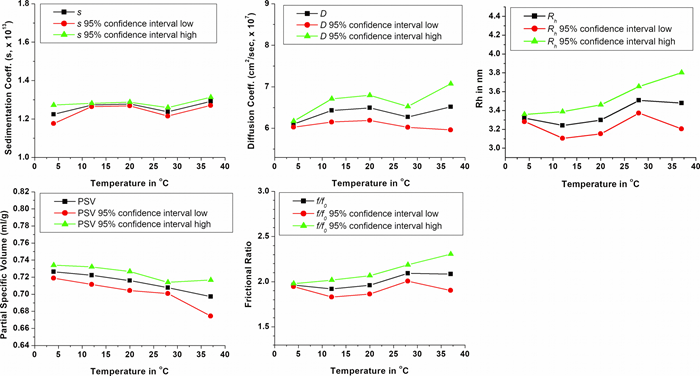 |
| Figura 2. Parámetros hidrodinámicos obtenidos del algoritmo genético - Resultados del análisis de Monte Carlo a partir de la superposición de muestras duplicadas a 4, 12, 20, 28 y 37 °C |
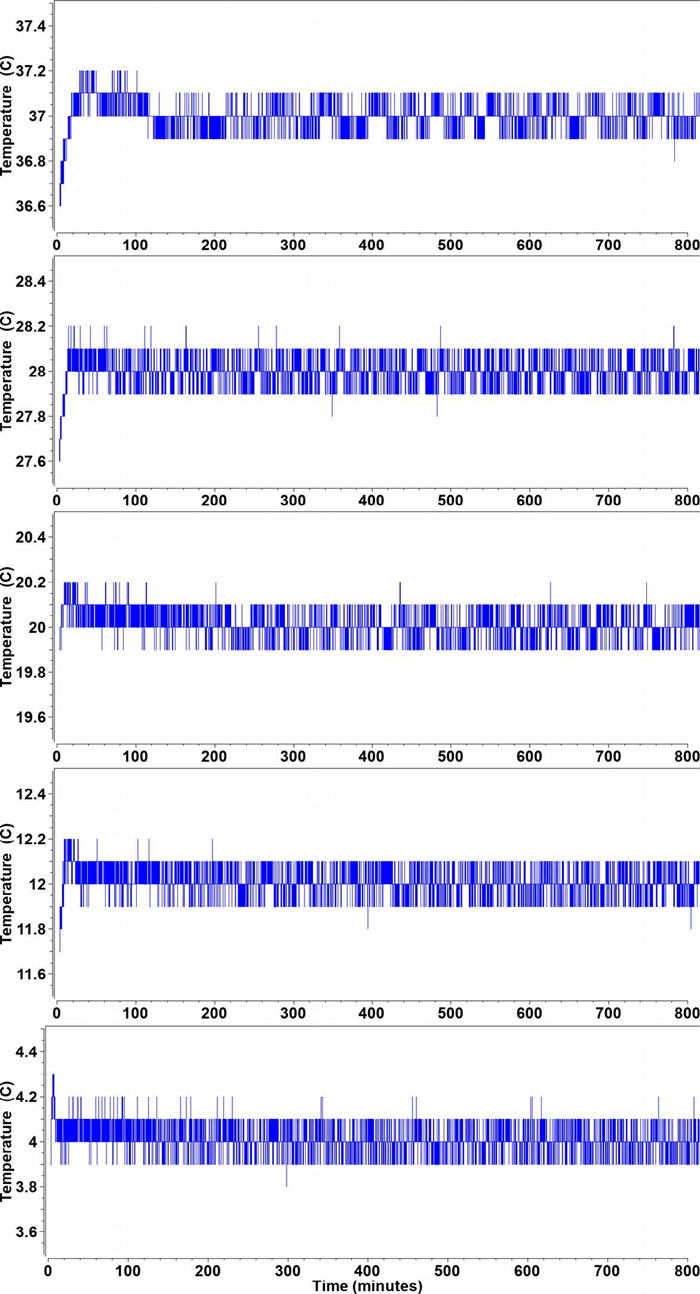 |
| Figura 3. Perfiles de temperatura del grafico de tiempo-estado de la Optima AUC durante los primeros 800 minutos obtenidos de cada experimento a una temperatura diferente en el intervalo de 4 a 37 °C. Excepto por un breve período de enfriamiento adiabático durante el estiramiento del rotor, la temperatura varía en raras ocasiones más de +/-0,1 °C a cualquier temperatura. La temperatura ambiente era de 24 °C. |
Reconocimientos
BD agradece el apoyo de la Fundación Nacional de Ciencias, subvención NSF-ACI-1339649, y de la NIH, subvención GM-120600, para el desarrollo del UltraScan Science Gateway y el software UltraScan. Este estudio contó con un mayor respaldo por parte de Beckman-Coulter. Las asignaciones de tiempo del superordenador se proporcionaron a través de la subvención TG-MCB070039 de la Fundación Nacional de Ciencias (a BD). Reconocemos el apoyo del Instituto del Cáncer de San Antonio a través de la subvención P30 CA054174 para apoyar al Centro de Ultracentrifugación Analítica de Conjuntos Macromoleculares en el Centro de Ciencias de la Salud de la Universidad de Texas en San Antonio.
Referencias
- English LR, Tischer A, Demeler A, Demeler B, Auton M, and ST Whitten. Conformational bias in disordered proteins correlates with mean hydrodynamic size. (submitted)
- Demeler B, Gorbet G, Zollars D, Dubbs B, Brookes E, Cao W. (2016) UltraScan-III version 3.5: A comprehensive data analysis software package for analytical ultracentrifugation experiments. http://www.ultrascan3.uthscsa.edu/.
- Demeler B., and G. Gorbet. Analytical Ultracentrifugation Data Analysis with UltraScan-III. Analytical Ultracentrifugation: Instrumentation, Software, and Applications. Eds: Uchiyama S., Stafford W. F. and T. Laue, Springer. 2016, Ch 8,119-143
- Kell, SG. Density, thermal expansivity, and compressibility of liquid water from 0.deg. to 150.deg.. Correlations and tables for atmospheric pressure and saturation reviewed and expressed on 1968 temperature scale. J. Chem. Eng. Data, 1975, 20 (1), pp 97–105
- Cao W. and B. Demeler. Modeling analytical ultracentrifugation experiments with an adaptive space-time finite element solution of the Lamm equation. (2005) Biophys J. 89(3):1589-602.
- Cao, W and B. Demeler. Modeling Analytical Ultracentrifugation Experiments with an Adaptive Space-Time Finite Element Solution for Multi-Component Reacting Systems. Biofímacos. J. (2008) 95(1):54-65
- Brookes, E. H., Cao W., and Demeler, B. (2010). A two-dimensional spectrum analysis for sedimentation velocity experiments of mixtures with heterogeneity in molecular weight and shape. Eur. Biofímacos. J. 39:405-414.
- Brookes, E. H. and Demeler, B. (2007). Parsimonious Regularization using Genetic Algorithms Applied to the Analysis of Analytical Ultracentrifugation Experiments. GECCO Proceedings ACM. 978-1-59593-697-4/07/0007.
- Demeler B. and Brookes E. Monte Carlo analysis of sedimentation experiments. Colloid Polym Sci (2008) 286(2) 129-137
- Brookes, E. H., and B. Demeler. 2008. Parallel computational techniques for the analysis of sedimentation velocity experiments in UltraScan. Colloid Polym. Sci. 286:138-148.
- Schuck P, Demeler B. Direct Sedimentation Boundary Analysis of Interference Optical Data in Analytical Ultracentrifugation. Biophysical Journal, 76:2288-2296 (1999)
- Demeler, B Methods for the Design and Analysis of Sedimentation Velocity and Sedimentation Equilibrium Experiments with Proteins. Cur. Protoc. Prot. Sci. (2010) Chapter 7:Unit 7.13.
CENT-3453APP01.18

